La molécule d’une protéine dispose d’un nombre astronomique de positionnement dans l’espace tridimensionnel. Ce pliage de protéine se fait dans un délai de perception très court et difficile à observer. Cette structure est essentielle à l’existence de la vie. Elle l’est aussi pour certaines maladies, qui dépendent de l’organisation maladroite de ces mêmes molécules.
Omique
Plusieurs expériences ont été menées pour étudier ce repliement qui questionne tant. Ces expériences font appel à la résonance magnétique nucléaire, la microscopie cryo-électronique. La prédiction de la structure du pliage de protéine est un des objectifs majeurs en matière de bio ingénierie. Par exemple, la conception de médicaments ou de nouveaux enzymes. Les enzymes sont utilisés par exemple dans l’industrie des détergents, les domaines de l’analyse et des capteurs. Mais c’est plus récemment dans les recherches ”Omiques” qu’ils ont fait leur apparition: des technologies apparues ces dernières années.
Ces recherches mêlent chimie, physique et informatique. Elles ont permis de développer des technologies qui mettent en œuvre une ingénierie d’analyse systématique du contenu du vivant à l’échelle moléculaire. Une plate-forme de services technologiques adaptés et fonctionnant à la demande peut désormais, à partir de n’importe quel échantillon contenant de la matière organique, réaliser une méta-analyse de type « omique ».
Le programme AlphaFold
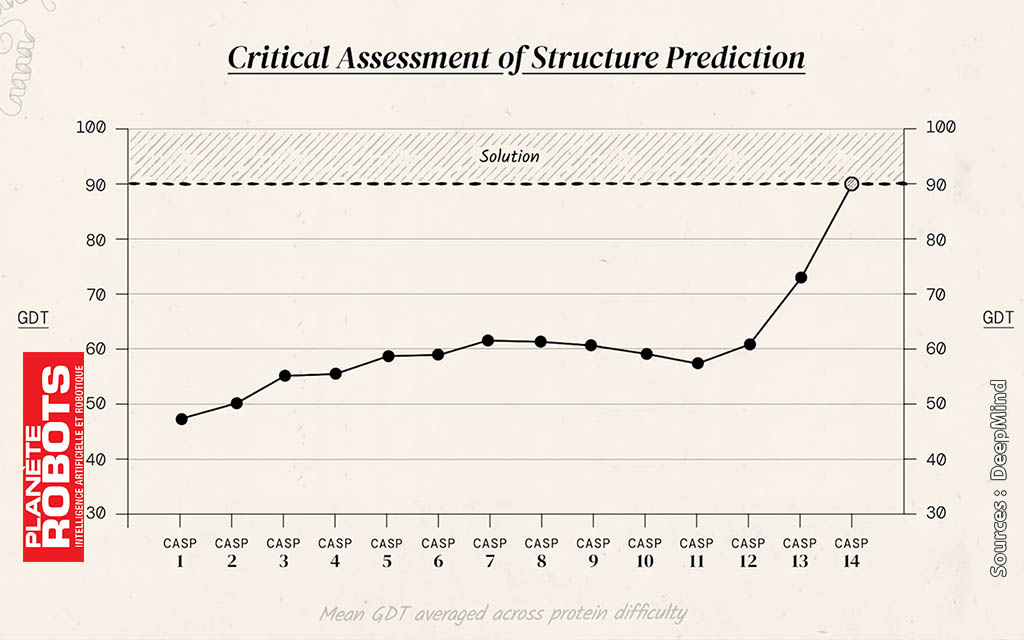
Pour arriver à percer ce mystère du repliement des protéines, l’équipe de DeepMind a utilisé le système CASP (évaluation critique de la prédiction de la structure des protéines). L’équipe de DeepMind a formé son IA sur 170 000 structures protéiques. Elles se basent sur la banque de données des protéines, ainsi que sur de grandes bases de données. Ces grandes bases, contiennent elles, des séquences protéiques de structure inconnue. Pour le matériel, ils ont utilisé 128 unités de traitement des tenseurs. Ces unités équivalent à entre 100 et 200 unités de traitement graphique (GPU). Ces unités ont calculé en quelques semaines. Selon l’équipe, c’est “une quantité de calcul relativement modeste. À noter que nous sommes dans un contexte de la plupart des grands modèles de pointe utilisés dans l’apprentissage machine”. Ainsi, AlphaFold est né, en tant que programme d’intelligence artificielle, développé par DeepMind de Google.
“En plus d’accélérer la compréhension des maladies connues par le programmes, il y a d’autres intérêts. Il est a noter, qu’il a enthousiasmé par le potentiel de ces techniques pour explorer les centaines de millions de protéines. Pour certaines d’entre-elles nous ne disposons pas encore de modèles. Cela représente pour les chercheurs un vaste terrain de biologie inconnu.” déclare l’équipe. “Parmi les protéines indéterminées, certaines pourraient avoir des fonctions nouvelles. Ces découvertes sont passionnantes. Tout comme un télescope nous aide à voir plus loin dans l’univers inconnu – des techniques comme AlphaFold pourraient nous aider à les trouver”.
source :
https://www.futuretimeline.net/blog/2020/11/30-protein-folding-artificial-intelligence.htm
Des robots origami injectés dans le corps qui viendraient bloquer la croissance d’une tumeur.



